|
Weitere Infos: |
|
Aktuelle Fachartikel: |
|
|
|
|
Wenn zwei sich binden |
|
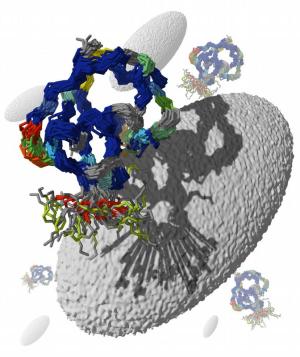
Proteine - die molekularen Maschinen unserer Zellen - besitzen einen entscheidenden Unterschied gegenüber technischen Maschinen, wie wir sie aus unserem Alltag kennen. Je nach Aufgabe, die sie erfüllen, können Proteine ihre Struktur innerhalb winzigster Bruchteile von Sekunden anpassen. Aber auch bei der Zusammenarbeit mit anderen Proteinen zählt die äußere Form. Anders als bisher angenommen, werden jedoch nicht alle Proteine zunächst von ihrem Partner passgenau zurechtgebogen. Es gibt Proteine, die ihre Struktur ständig ändern und dabei auch ganz ohne fremde Hilfe "in Form" kommen. Dies hat ein Wissenschaftler-Team vom Göttinger Max-Planck-Institut für biophysikalische Chemie herausgefunden, indem sie einen wahren Verwandlungskünstler unter den Proteinen untersuchten - das Ubiquitin. Den Forschern gelang es erstmals, Bewegungen des Proteins im Bereich von milliardstel bis millionstel Sekunden sichtbar zu machen. |
|
Molekulare Maschinen sind die Hauptakteure bei der Umsetzung lebenswichtiger Prozesse in unserem Körper. Ihre komplexen Aufgaben, etwa als Enzyme, Transport- oder Signalmoleküle, können sie aber nur dann erfüllen, wenn ihre Bausteine - eine oder mehrere Aminosäureketten - sich nach der Produktion in die richtige Struktur falten. Die Proteinstruktur ist alles andere als starr und ändert sich bereits in winzigen Bruchteilen von Sekunden. Nur so können Proteine, meist in Zusammenarbeit mit anderen Proteinen, ihre komplexen Aufgaben in der Zelle erfüllen. Perfekt wie Schlüssel und Schloss passen Proteine allerdings nur selten zusammen. Vielmehr gilt: Was nicht passt, wird passend gemacht. Das Protein wird vom Partner regelrecht in Form gebogen. Doch gilt dies für alle Proteine, die miteinander wechselwirken? Zunehmend diskutieren Wissenschaftler auch ein anderes Modell. Proteine könnten durch ständige Bewegungen das benötigte Repertoire an Passformen auch zufällig erzeugen. Wie ein Schlüsselbund, der für jedes Schloss den passenden Schlüssel parat hat, steckten so die Passformen bereits im Protein selbst. Passt eine dieser Formen dann zufällig zu einem seiner Partner, so binden die beiden Proteine aneinander. Doch wie lassen sich solche Bewegungen beobachten? Schnappschüsse von Proteinbewegungen im Nano- bis Mikrosekundenbereich Damit sich die Form eines Proteins ändert, müssen sich seine molekularen "Zahnräder", Molekülgruppen und Aminosäuren, entsprechend bewegen. Messen konnten Wissenschaftler bisher Bewegungen, die innerhalb von Nanosekunden (milliardstel Sekunden) oder sogar noch schneller ablaufen, sowie langsamere Bewegungen im Mikrosekunden- bis Millisekundenbereich. Viele Proteinbewegungen, ebenso wie viele biochemische Prozesse, laufen aber gerade im Bereich zwischen Nano- und Mikrosekunden ab - ein Bereich, der bisher mit keiner Methode beobachtbar war. Einem Team von Strukturbiologen und Biophysikern des Max-Planck-Instituts für biophysikalische Chemie gelang es nun, diese "Beobachtungslücke" zu schließen. Erstmals konnten die Wissenschaftler Bewegungen einzelner Zahnräder eines Proteins in diesem bisher unzugänglichen Zeitfenster sichtbar machen. Als Untersuchungsobjekt diente den Forschern das Ubiquitin, ein wahrer Verwandlungskünstler unter den Proteinen, das nicht nur sein Äußeres, sondern auch seine Partner häufig wechselt. Ubiquitin ist Teil eines ausgeklügelten Recycling-Systems, das beschädigte oder ausgediente Proteine in ihre Bestandteile zerlegt und wiederverwertet. Als eine Art Aufkleber markiert es Proteine als zellulären "Müll". Derart markierte Proteine werden dann im zentralen "Müllschredder" der Zelle - dem Proteasom - in ihre Bausteine zerhackt. Bereits 46 verschiedene Strukturen von Ubiquitin sind bekannt, in 21 dieser Strukturen bindet Ubiquitin an jeweils andere Partner. Die spannende Frage beantworten diese strukturellen Momentaufnahmen allerdings nicht: Verändert Ubiquitin sein Äußeres spontan, oder wird ihm die Form von seinem jeweiligen Bindungspartner aufgezwungen? Um diese Frage zu beantworten, kombinierten die Wissenschaftler um Bert de Groot, Christian Griesinger und Helmut Grubmüller eine Fülle von Kernspinresonanzspektroskopie-Experimenten mit Molekulardynamik-Simulationen. Durch trickreichen Einsatz verschiedener Lösungsmedien, die sich wie schwache Flüssigkristalle verhalten und dadurch dem Ubiquitin eine Vorzugsorientierung aufzwingen, konnten sie die Bewegung von Ubiquitin gewissermaßen aus verschiedenen Blickwinkeln wie mit Schnappschüssen festhalten. Mit Moleküldynamiksimulationen ließ sich daraus anschließend die Bewegung rekonstruieren. "Wir sehen, dass alle 46 bekannten Strukturen des Ubiquitins auch schon in Lösung vorhanden sind, so als ob Ubiquitin seine Bindungspartner erwarte und vorauseilend die richtige Bindungskonformation einnähme", erläutert Christian Griesinger. Dabei bewegt sich nicht das ganze Protein. "Bewegung sehen wir vor allem an den Kontaktstellen, mit denen es an andere Proteine bindet", so Griesinger. Neuer Mechanismus für molekulare Erkennung Zumindest für Ubiquitin muss das Lehrbuch-Kapitel über Wechselwirkungen zwischen Proteinen nun umgeschrieben werden. Dass es ständig sein Äußeres ändert, kann leicht erklären, warum es zu einer Vielzahl unterschiedlichster Proteine passt. Eine Ausnahme unter den Proteinen sei das Ubiquitin damit aber vermutlich nicht, sagt Bert de Groot. Die Forscher vermuten, dass neben dem Ubiquitin auch weitere Proteine spontan ihre Form ändern. Es könne ein ganz generelles Modell für molekulare Erkennung sein, so de Groot. Lediglich trockener Lehrbuchstoff sind die neuen Erkenntnisse der Forscher jedoch keineswegs. Wechselwirkungen von Proteinen spielen bei zahlreichen Prozessen der Zelle eine entscheidende Rolle, unter anderem bei der Signal-Weiterleitung zwischen Zellen. Ist diese gestört, weil bestimmte Proteine nicht mehr zusammenarbeiten, sind die Folgen oft fatal. Die neuen Methoden der Forscher könnten dazu beitragen, zumindest einige dieser Störungen besser zu verstehen. Auch die Wirkung vieler Arzneimittel beruht auf Wechselwirkungen zwischen Proteinen oder Proteinen mit anderen Molekülen. Die Wissenschaftler untersuchen derzeit, ob die neuen Erkenntnisse genutzt werden können, Medikamente in ihrer Wirksamkeit noch spezifischer zu machen. |
|
|
|
|
Suche nach themenverwandten Internetseiten: |
|
|