|
|
|
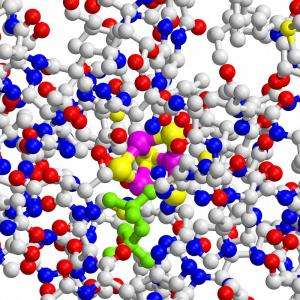
Das IspH-Protein von Bakterien verfügt über einen ungewöhnlichen Eisen-Schwefel-Cluster (lila/gelb), der die Bildung lebenswichtiger Bausteine katalysiert. Dieses Zentrum könnte der ideale Angriffspunkt für neue Antibiotika werden.
[Bildquelle: Prof. Dr. Michael Groll, TUM]
|
Die lebenswichtigen Naturstoffe aus der Terpen- und Steroidklasse stellen die Zellen fast aller Organismen aus den kleinen Isoprenbausteinen Dimethylallylpyrophosphat (DMAPP) und Isopentenylpyrophosphat (IPP) her. Säugetiere und eine große Zahl weiterer Organismen bauen diese über den so genannten Mevalonat-Weg auf. Fast alle krankmachenden Bakterien, so auch der Malariaerreger Plasmodium falciparum, haben dagegen eine andere Möglichkeit gefunden, diese wichtigen Substanzen herzustellen. Ihr Sonderweg könnte den Bakterien nun zum Verhängnis werden: Die TUM-Wissenschaftler haben die strukturellen Grundlagen des letzten Schritts der bakteriellen Isopren-Synthese aufgeklärt. Das entscheidende Enzym verfügt über eine äußerst ungewöhnliche Struktur, ähnlich der eines dreiblättrigen Kleeblatts. Es könnte sich als entscheidender Angriffspunkt für neue, maßgeschneiderter Antibiotika erweisen. Die Arbeiten zur Aufklärung der bakteriellen Isoprenbaustein-Produktion wurden bereits vor zwölf Jahren am Lehrstuhl für Organische Chemie und Biochemie durch Professor Adelbert Bacher in Zusammenarbeit mit den Privatdozenten Wolfgang Eisenreich und Felix Rohdich begonnen. Im Laufe der Jahre entdeckte das Team die meisten Reaktionsschritte des neuen Stoffwechselwegs. Der letzte vom Enzym IspH katalysierte Schritt entzog sich jedoch hartnäckig der strukturellen Aufklärung. Die früheren Messungen legten nahe, dass das aktive Zentrum ein Eisen-Schwefel-Cluster mit drei Eisen und vier Schwefelatomen sein müsste. Andere Wissenschaftler zweifelten die Ergebnisse an, und jahrelang gelang es nicht, eine Kristallstruktur des Enzyms zu bestimmen, die den Beweis hätte liefern können. Größtes Problem dabei war die Sauerstoffempfindlichkeit des Enzyms, das an der Luft schon in kürzester Zeit degeneriert und dabei Struktur und Funktion einbüßt. Erst kürzlich gelang es einer Arbeitsgruppe von der Justus-Liebig-Universität in Gießen, die Röntgenkristallstruktur der offenen Form des Enzyms zu entschlüsseln. Doch diese Struktur lieferte kaum Informationen zum Ablauf der vom Enzym katalysierten Umsetzung. Das Forscherteam um Professor Groll, Dr. Eppinger und Dr. Gräwert gelang es nun, auch die Röntgenkristallstruktur der geschlossenen Form zu lösen, die die genaue Faltung der Proteinkette und die chemische Umgebung des aktiven Zentrums zeigt. Nun konnten sie durch Computersimulationen und Mutagenese-Experimente, bei denen Escherichia coli-Bakterien dazu gebracht wurden fehlerhafte IspH-Enzyme zu synthetisieren, den Mechanismus der Reaktion detailliert untersuchen. Röntgenkristallstruktur, kinetische Messungen und Mutagenesestudien bestätigten schließlich die schon vor Jahren vorgeschlagene, ungewöhnliche Anordnung von drei Eisen und vier Schwefelatomen im aktiven Zentrum des Enzyms. "Nachdem nun Ort, chemischer Ablauf und beteiligte Partner der IspH-Reaktion bekannt sind," erläutert Groll, "besteht ein neuer Ansatzpunkt gezielt Substanzen zu entwickeln, die den letzten Schritt der bakteriellen Synthese von Isoprenbausteinen blockieren und Erreger gezielt abtöten könnten. Da Enzym und Reaktion in Säugetieren nicht vorkommen, sollten diese Verbindungen für Menschen keine oder nur geringe Nebenwirkungen besitzen." Die Arbeiten wurden unterstützt von der Hans-Fischer-Gesellschaft und dem Stifterverband für die Deutsche Wissenschaft (Projekt-Nr. 11047: Forschungsdozentur Molekulare Katalyse).
|