Warum Enzyme, die zur technischen Herstellung von Wasserstoff genutzt werden, so empfindlich gegenüber Sauerstoff sind, berichten Wissenschaftler der RUB-Fakultät für Biologie und Biotechnologie im Journal of Biological Chemistry.
Gemeinsam mit Berliner Forschern untersuchten sie mit spektroskopischen Methoden den Zeitverlauf der Vorgänge, die zur Inaktivierung der Enzymfunktion in so genannten Hydrogenasen führen.
"Hydrogenasen könnten von herausragender Bedeutung sein, um Wasserstoff mit Hilfe von biologischen oder chemischen Katalysatoren zu gewinnen", erklärt Camilla Lambertz aus der RUB-Arbeitsgruppe Photobiotechnologie.
"Ihre hohe Sauerstoffempfindlichkeit stellt allerdings ein großes Problem dar. Unsere Ergebnisse können helfen, in Zukunft robustere Enzyme zu entwickeln."
Sauerstoff als Freund und Feind
Für die meisten Tiere und Pflanzen ist Sauerstoff überlebensnotwendig, doch wirkt er in zu hohen Konzentrationen auf viele Lebewesen giftig und einige Organismen können sogar nur ganz ohne Sauerstoff existieren. Die Sensibilität gegenüber Sauerstoff findet man auch auf der Ebene der Proteine. So ist eine Vielzahl von Enzymen, z.B. Hydrogenasen, bekannt, die durch Sauerstoff irreversibel zerstört werden. Hydrogenasen sind biologische Katalysatoren, die Protonen und Elektronen in technisch nutzbaren Wasserstoff umwandeln. Das RUB-Team um Prof. Dr. Thomas Happe erforscht sogenannte [FeFe]-Hydrogenasen, die besonders viel Wasserstoff produzieren können. Die Bildung von Wasserstoff findet u.a. an zwei speziellen Eisenatomen des Enzyms statt, die gemeinsam mit anderen Atomen das reaktive Zentrum formen.
Sauerstoff greift Eisenzentren an
Gemeinsam mit der Arbeitsgruppe von Dr. Michael Haumann in Berlin fanden die Forscher heraus, dass Sauerstoff sich an das Zweieisenzentrum der Hydrogenase bindet und dadurch einen anderen Bereich des Enzyms aus vier weiteren Eisenatomen ausschaltet. In dem vom BMBF geförderten Forschungsprojekt zeigten sie erstmals die verschiedenen Phasen des Inaktivierungsprozesses mit der so genannten Röntgenabsorptionsspektroskopie. Für die spezielle Art der Messung nutzen die Wissenschaftler die Synchrotronstrahlungsquelle Swiss Light Source in der Schweiz, die besonders starke Röntgenstrahlen erzeugt und somit die Charakterisierung von Metallzentren in Proteinen erlaubt. So bestimmten die Forscher unter anderem die chemische Natur der Eisenzentren und den Abstand zu den Nachbaratomen mit atomarer Auflösung.
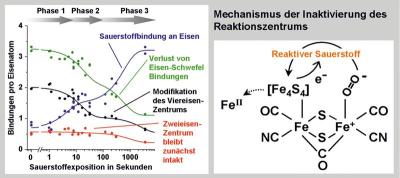
Inaktivierung in drei Stufen
Das Team der Bochumer und Berliner Wissenschaftler setzte ein neues experimentelles Protokoll ein. Sie brachten die Hydrogenaseproben erst für einige Sekunden bis Minuten und zuletzt für einige Stunden mit Sauerstoff in Kontakt und unterbanden dann alle weiteren Reaktionen durch Tiefgefrieren in flüssigem Stickstoff. Aus den anschließend gewonnenen spektroskopischen Daten erstellten sie ein Modell für einen dreistufigen Prozess der Inaktivierung. Laut Modell bindet zunächst ein Sauerstoffmolekül an das Zweieisenzentrum der Hydrogenase, woraufhin eine aggressive Sauerstoffspezies entsteht. Diese greift in der folgenden Phase das Viereisenzentrum an und modifiziert es. Im letzten Schritt binden weitere Sauerstoffmoleküle und der gesamte Komplex zerfällt. "Der ganze Vorgang beinhaltet also mehrere aufeinanderfolgende Reaktionen, die zeitlich klar getrennt auftreten", so Lambertz. "Die Geschwindigkeit des Gesamtprozesses wird möglicherweise durch den Schritt bestimmt, in dem die aggressive Sauerstoffspezies vom Zwei- zum Viereisenzentrum wandert. Wir bereiten gerade weitere Experimente vor, um das zu prüfen."
Zusatzinformationen:
Camilla Lambertz, Nils Leidel, Kajsa G. V. Havelius, Jens Noth, Petko Chernev, Martin Winkler, Thomas Happe and Michael Haumann:
O2-reactions at the six-iron activesite (H-cluster) in [FeFe]-hydrogenase.
In: Journal of Biological Chemistry; online veröffentlicht am 19. September 2011, DOI 10.1074/jbc.M111.283648
Quelle: Ruhr-Universität Bochum, RUB
Aktualisiert am 12.10.2011.
Permalink: https://www.internetchemie.info/news/2011/oct11/hydrogenase-inaktivierung.php
© 1996 - 2026 Internetchemie ChemLin