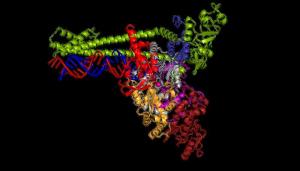
Wenn sich ein Mensch mit dem neuartigen Coronavirus SARS-CoV-2 infiziert, vermehrt sich der Erreger in dessen Zellen rasend schnell. Dazu muss das Virus sein Erbgut, das aus einem langen RNA-Strang besteht, vervielfältigen. Diese Aufgabe übernimmt die virale "Kopiermaschine", Polymerase genannt. Wissenschaftler um Patrick Cramer vom Göttinger Max-Planck-Institut für biophysikalische Chemie haben jetzt die räumliche Struktur der Corona-Polymerase entschlüsselt. Damit lässt sich erforschen, wie antivirale Substanzen wirken, die die Polymerase blockieren. Eine von ihnen ist der Hoffnungsträger Remdesivir. Außerdem können neue Wirkstoffkandidaten gesucht werden.
"Im Angesicht der derzeitigen Pandemie wollten wir helfen", sagt Max-Planck-Direktor Cramer. "Wir verfügen über umfassende Erfahrung, Polymerasen zu untersuchen." So lag das Forschungsthema für die Wissenschaftler auf der Hand.
"Das Überraschendste für uns war, dass der Aufbau der Coronavirus-Kopiermaschine aus der Reihe fällt, denn sie unterscheidet sich von anderen Polymerase-Strukturen", erklärt Hauke Hillen. Die Corona-Polymerase bindet zwar so an die RNA, wie es auch von anderen Virusarten bekannt ist. Doch diese Polymerase besitzt ein weiteres Element, mit der sie sich an der RNA festklammert, bis sie das Erbgut kopiert hat. Das ist gerade für das Coronavirus wichtig, denn sein Erbgut besteht aus rund 30.000 Bausteinen und ist damit besonders lang, das Kopieren eine echte Mammutaufgabe.
Im Detail verstehen, wie antivirale Substanzen wirken
Zu wissen, wie die Coronavirus-Polymerase Atom für Atom aufgebaut ist, eröffnet neue Möglichkeiten, den Erreger besser zu verstehen und zu bekämpfen. Im nächsten Schritt will das Forscherteam um Cramer im Detail untersuchen, wie antivirale Substanzen die Vermehrung von Coronaviren blockieren. "Auf Remdesivir, das die Corona-Polymerase direkt blockiert, ruhen viele Hoffnungen. Durch die Polymerase-Struktur könnte es möglich werden, bereits vorhandene Substanzen wie Remdesivir zu optimieren und ihre Wirkung zu verbessern. Doch wir wollen auch nach neuen Substanzen fahnden, die die Virus-Polymerase stoppen können", so Cramer.
Ihre Ergebnisse haben die Göttinger Forscher in einem Manuskript bereits im Internet veröffentlicht. "Wir wollten unsere Erkenntnisse sofort mit der internationalen Forschungsgemeinschaft teilen, denn es muss jetzt, wo wir uns mitten in der Pandemie befinden, besonders schnell gehen," berichtet Lucas Farnung, der in Kürze auf eine Professur an die US-amerikanische Harvard University wechseln wird.
Die Corona-Kopiermaschine 100.000-fach vergrößert
Der Weg hin zur dreidimensionalen Struktur der Corona-Polymerase war steinig. "Zunächst mussten wir die Polymerase aus drei gereinigten Proteinen im Reagenzglas nachbauen. Nach einigen Optimierungen war sie schließlich funktionsfähig", erläutert Goran Kokic. "Nur so konnten wir untersuchen, wie sie arbeitet." Der Wissenschaftler hatte dazu auf die Schnelle einen speziellen Test etabliert, um die Aktivität der Polymerase bestimmen zu können.
Im Anschluss untersuchte das Team die Proben im Elektronenmikroskop bei über 100.000-facher Vergrößerung - und zunächst machte sich Enttäuschung breit: "Obwohl wir zehn Tage und Nächte rund um die Uhr Bilder aufgenommen hatten, konnten wir keine detaillierten Einblicke in die Struktur gewinnen", erinnert sich Christian Dienemann, Experte für Elektronenmikroskopie. "Allerdings sah eine Probe anders aus, irgendwie seltsam. Unser erster Gedanke war, sie zu verwerfen. Glücklicherweise haben wir das nicht getan: Ausgerechnet diese Probe hat uns die hochwertigen Daten geliefert, die wir unbedingt brauchten," erzählt Dimitry Tegunov, Datenverarbeitungsexperte der Gruppe, der auch die Software programmiert hat, um schnell große Mengen von Bilddaten zu verarbeiten.
Die Entschlüsselung der Polymerase-Struktur soll nicht der letzte Beitrag der Göttinger Forscher zur Bewältigung der Pandemie sein: "Wir haben auch die sogenannten Helferfaktoren im Visier, die die Virus-RNA so verändern, dass sie durch das menschliche Immunsystem nicht abgebaut werden kann", so Cramer. "Und natürlich hoffen wir als Strukturbiologen, weitere Angriffspunkte im Virus zu finden, die mittelfristig neue Therapiestrategien eröffnen."
Zusatzinformationen:
Hauke S. Hillen, Goran Kokic, Lucas Farnung, Christian Dienemann, Dimitry Tegunov und Patrick Cramer:
Structure of replicating SARS-CoV-2 polymerase.
In: Preprint bioRxiv; Vorabveröffentlichung vom 27. April 2020, DOI 10.1101/2020.04.27.063180
Quelle: Max-Planck-Institut für biophysikalische Chemie, Göttingen
Aktualisiert am 28.04.2020.
Permalink: https://www.internetchemie.info/news/2020/apr20/räumliche_struktur_der_corona_polymerase.php
© 1996 - 2026 Internetchemie ChemLin